A leishmaniose tegumentar americana é uma dermatose infecciosa causada por protozoários do gênero Leishmania e compreende amplo espectro de manifestações clínicas dependente das espécies do parasita envolvidas nas infecções e da resposta imunogenética do hospedeiro. O uso de técnicas de amplificação de DNA do parasita baseadas em reação em cadeia da polimerase (PCR) e a recente aplicação de técnicas acopladas, como a de dissociação de DNA em alta resolução, têm sido descritos como opção viável para a detecção e a identificação de Leishmania spp. em amostras biológicas.
ObjetivosIdentificar as espécies de Leishmania pela técnica de PCR‐alta resolução em biópsias de pele de pacientes atendidos em hospital e comparar com resultados obtidos por outras técnicas de identificação molecular.
MétodosEstudo retrospectivo que avaliou pacientes com suspeita de leishmaniose tegumentar americana atendidos em hospital da cidade de São Paulo. Biópsias parafinadas de 22 pacientes foram analisadas por PCR‐alta resolução para confirmação do diagnóstico e identificação das espécies de Leishmania.
ResultadosDos 22 pacientes com suspeita de leishmaniose tegumentar americana, 14 tiveram o parasita identificado: cinco casos (35,6%) de infecção por Leishmania amazonensis, quatro (28,5%) por Leishmania braziliensis, dois (14,4%) por L. amazonensis + Leishmania infantum chagasi, dois (14,4%) por Leishmania guyanensis e um caso (7,1%) por L. infantum chagasi. Em uma das amostras em que a presença de formas amastigotas foi confirmada em exame histopatológico, a técnica de PCR‐alta resolução não detectou o DNA do parasita.
Limitações do estudoEstudo retrospectivo com pequeno número de pacientes.
ConclusõesO método detectou e identificou espécies de Leishmania em blocos parafinados com sensibilidade de 96,4%, indicou a possibilidade de uso dessa metodologia na rotina diagnóstica.
A leishmaniose é uma doença infectoparasitária crônica causada por diversas espécies de protozoários do gênero Leishmania. No Brasil, as espécies envolvidas nas infecções que determinam as manifestações tegumentares são principalmente Leishmania (Viannia) braziliensis e Leishmania (Leishmania) amazonensis, seguidas das espécies Leishmania (Viannia) guyanensis, Leishmania (Viannia) lainsoni, Leishmania (Viannia) shawi, Leishmania (Viannia) naiffi e Leishmania (Viannia)linderbergi, com menor incidência.1–3 Mais recentemente têm sido relatados casos isolados de leishmaniose tegumentar americana (LTA) causadas por Leishmania (Leishmania) infantum chagasi em pacientes com ou sem coinfecção por HIV, nas regiões Centro‐Oeste e Sudeste do país.4,5
Na pele, a apresentação clínica inicial mais comum da LTA é de úlcera emoldurada no local da inoculação do parasita pelo inseto. Nessa forma é possível encontrar o parasita por pesquisa direta do esfregaço do fundo de úlceras recentes. No entanto, há outras formas de manifestações cutâneas, como verrucosa, papulosa, nodular, sarcoídica, placa infiltrada e vegetante, nas quais a presença do parasita no tecido é rara e a histopatologia é inespecífica, varia de infiltrado inflamatório linfoplasmocitário até granuloma com ou sem necrose tecidual, dificulta assim o diagnóstico diferencial com outras doenças infecciosas, inflamatórias e até neoplásicas.6–9 A imuno‐histoquímica tecidual com anticorpos antileishmania contribui para o diagnóstico, mas apresenta sensibilidade por volta de 66% dos casos. A reação intradérmica de Montenegro positiva (˃ 5mm) pode auxiliar por ter alto valor preditivo; no entanto, esse exame apresenta algumas limitações: é negativo nos primeiros meses de infecção e em pacientes anérgicos e mantém‐se positivo mesmo após a cura da doença.1,6
Assim, a leishmaniose pode ser diagnosticada de várias maneiras, todas com vantagens e limitações. No entanto, não há um exame padrão ouro para diagnosticar a doença. A microscopia é útil em áreas endêmicas, mas apresenta baixa sensibilidade e depende da avaliação de um patologista experiente, além de não ser possível a identificação das espécies do parasita. Os testes sorológicos podem fornecer um diagnóstico rápido, de fácil interpretação e são relativamente baratos; no entanto, há variações na sensibilidade e na especificidade nas diferentes regiões endêmicas, além da impossibilidade de identificação das espécies causadoras da doença.1,3,6
Diferentes métodos com abordagens moleculares que exploram características específicas do DNA de parasitas têm sido testados para o diagnóstico das leishmanioses. Inicialmente, as análises de padrões de fragmentos gerados por enzimas de restrição (RFLP) constituíam o método empregando DNA como alvo na identificação de Leishmania e de outros tripanossomatídeos.10–12 Outro tipo de análise de genótipos consiste na construção de sondas usadas em testes de hibridação molecular. Essa técnica foi empregada tanto em estudos discriminatórios entre espécies fortemente relacionadas quanto para o diagnóstico de infecções.13,14 A técnica de amplificação aleatória de DNA polimórfico (RAPD), que amplifica por reação em cadeia da polimerase (PCR) fragmentos com iniciadores aleatórios, também foi uma técnica adotada para analisar padrões específicos capazes de discriminar os organismos do gênero Leishmania.15,16 Em função de inúmeras vantagens, os testes baseados na técnica de PCR são as principais ferramentas usadas na detecção e identificação de Leishmania. Essa metodologia torna possível, além do uso de ínfimas quantidades de material biológico, a discriminação específica de organismos pela análise de polimorfismos dos alvos. Diferentes técnicas baseadas em PCR têm sido empregadas na identificação de Leishmania por apresentar vantagens como rapidez e sensibilidade e especificidade altas, quando comparadas às técnicas convencionais baseadas em microscopia e cultura de células.17
Diversas sequências de DNA têm sido usadas como alvo para a identificação específica de Leishmania, como o DNA do cinetoplasto (kDNA),18 o DNA ribossômico (rDNA),13,14 o gene g6pd, entre outros alvos.19,20 O kDNA compõe cerca de 15% do DNA do parasita e é formado por uma rede de moléculas circulares concatenadas, os maxicírculos e os minicírculos. Por organismo, são encontrados entre 10 mil e 20 mil minicírculos com tamanho de 0,5 a 2,5kb e cerca de 50 maxicírculos de 20 a 35kb que codificam proteínas mitocondriais e RNA ribossômico (rRNA) da organela.21,22 Entre os alvos descritos na literatura, o kDNA está entre os mais usados no diagnóstico molecular. O elevado número de cópias do alvo por genoma garante alta sensibilidade e constitui a principal vantagem da técnica. Porém, a acurácia dos ensaios pode ser afetada pela considerável heterogeneidade das sequências de kDNA.23
A caracterização de cistrons ribossômicos de diversos tripanossomatídeos viabilizou a descrição de sondas capazes de identificar alguns organismos. A princípio foi demonstrada a existência de polimorfismo nas sequências espaçadoras de genes ribossômicos em Trypanosoma.11 A caracterização de cistrons ribossômicos de seis espécies de Leishmania mostrou um padrão característico dessas espécies pontuado pela existência de um sítio de restrição reconhecido pela enzima PvuII. Esse sítio, presente na sequência codificadora do rRNA 18S ou SSU, foi explorado na construção de oligonucleotídeos capazes de identificar o gênero.13 Com base em polimorfismos na sequência desse mesmo gene, foram descritas sondas que puderam ser usadas na identificação de diferentes grupos de Leishmania.14 O sequenciamento de amplicons desse segmento gênico tem sido empregado na identificação de Leishmania. Dois pontos de mutação são encontrados nas posições 1714 e 1721da sequência SSU rDNA, capazes de discriminar (Leishmania) amazonensis e L. (Viannia) spp. Baseados nessa estratégia, diversos trabalhos que exploraram as características polimórficas do SSU rDNA foram feitos com abordagens distintas, focaram aspectos clínicos, epidemiológicos, ecológicos, entre outros.24–30
Entre as técnicas de identificação molecular mais usadas, destacamos as análises por eletroforese de isoenzimas, também denominadas zimodemas. A glicose‐6‐fosfato desidrogenase (G6PD) é uma das principais enzimas usadas na identificação de Leishmania por análise de zimodemas. O polimorfismo na estrutura dessas proteínas é reflexo de polimorfismos em nível gênico, o que tornou possível o emprego de marcadores moleculares capazes de discriminar e quantificar Leishmania por PCR convencional e em tempo real.19,20
Além desses, outros alvos de DNA descritos na literatura, como os internal transcribed spacers (ITSs) presentes no cistron de rDNA, o gene que codifica a proteína de choque térmico, hsp70, o gene cpb (cysteine proteinase), entre outros, têm sido explorados em testes de identificação de Leishmania, com base nas características polimórficas do DNA.17
O uso simultâneo de alvos diferentes também tem sido aplicado em reações de PCR convencionais com o objetivo de discriminar os patógenos das leishmanioses. A combinação de oligonucleotídeos desenhados a partir da sequência do gene que codifica o miniéxon adicionado por trans‐splicing a todos os RNA mensageiros de tripanosomatídeos, o SL RNA, foi descrita como capaz de discriminar, em uma única reação de PCR, as espécies causadoras da forma visceral da doença em um grupo e as espécies responsáveis pela forma tegumentar da doença em outros dois grupos, distintos por incluírem espécies dos subgêneros L. (Leishmania) ou L. (Viannia).31,32
Recentemente, foi descrito um importante polimorfismo no gene que codifica a proteína de choque térmico, HSP70, que associado à análise pela aplicação de análises de HRM (High Resolution Melting), método capaz de detectar diferenças na composição de nucleotídeos de produtos específicos de PCR, permite discriminar todas as espécies que circulam no território nacional e as espécies mais importantes encontradas no Mediterrâneo, na Índia e no norte da África.33 A aplicação dessa técnica tem se revelado útil no diagnóstico e na identificação de Leishmania em diferentes amostras, tanto de pacientes (em biópsias a fresco, como em material parafinado) quanto em animais reservatórios, como cães, e em hospedeiros flebotomíneos.
Testes diagnósticos que envolvem a detecção de ácidos nucleicos de parasitas, principalmente aqueles baseados na amplificação específica de DNA, como as PCR, são descritos como opções altamente sensíveis e específicas, com capacidade potencial de quantificar e identificar as espécies infectantes.8,34–36 Análises de HRM feitas no fim de reações de PCR em tempo real são capazes de detectar diferenças termodinâmicas no perfil de dissociação de amplicons, determinam assinaturas específicas como resultado de pequenas diferenças na composição nucleotídica das espécies. A técnica de PCR‐HRM tem sido usada na identificação não só de espécies de Leishmania, mas também de outros agentes infecciosos, e tem sido descrita como uma ferramenta relativamente barata, rápida e eficaz para detecção e discriminação das espécies de Leishmania em todas as regiões endêmicas do mundo.34,37,38
MétodoO estudo retrospectivo foi realizado na Clínica de Dermatologia de um hospital terciário de São Paulo, analisou os dados de 22 pacientes com diagnóstico de LTA atendidos de janeiro de 2005 a dezembro de 2018. O presente estudo foi aprovado pelo Comitê de Ética (CAAE: 48719115.0.0000.5479) e os dados clínicos, epidemiológicos e resultados de exames complementares foram obtidos em análise de prontuário.
A Leishmania sp. foi identificada com a aplicação da metodologia PCR‐HRM, como previamente descrito por nosso grupo,33 a partir de amostra de DNA purificado dos blocos de parafina obtidos do laboratório de patologia do hospital. O envio das amostras obedeceu a um critério de teste cego, inclusive nos casos em que mais de uma amostra foi obtida do mesmo paciente. As biópsias parafinadas passaram por um processo de remoção de parafina com sucessivos banhos de xilol aquecido, seguidos de banhos de etanol e posterior hidratação da amostra. O DNA das biópsias foi então extraído e purificado com o uso do kit DNeasy Blood & Tissue (Qiagen), de acordo com o protocolo sugerido pelo fabricante. Uma primeira etapa, denominada pré‐amplificação, foi feita com oligonucleotídeos iniciadores baseados na sequência do gene hsp70 de Leishmania que amplifica, por PCR convencional, um segmento do DNA que contém sítios polimórficos passíveis de serem analisados por HRM. Os produtos da pré‐amplificação foram então submetidos a reações de PCR em tempo real, com o uso do kit MeltDoc Master Mix (Life Technologies), em que três regiões distintas do gene foram amplificadas independentemente. As análises de HRM foram feitas no fim de cada reação de PCR em tempo real e os perfis de dissociação de DNA foram gerados pelo software High Resolution Melting v. 3.0.1 (Life Technologies) e comparados aos perfis gerados a partir de amostras de DNA de cepas referência de Leishmania spp. [L. (L.)infantum chagasi (MCER/BR/1981/M6445), L.(L.) major MHOM/IL/81/Friedlin), L. (L.)amazonensis (MHOM/BR/1973/M2269), L.(L.) mexicana (MNYC/BZ/62/M379), L. (L.) lainsoni (MHOM/BR/81/M6426), L. (V.)braziliensis (MHOM/BR/1975/M2903), L. (V.)guyanensis (MHOM/BR/1975/M4147), L. (V.)naiffi (MDAS/BR/1979/M5533) e L. (V.)shawi (MCEB/BR/84/M8408)].
Além disso, os resultados obtidos por PCR‐HRM foram comparados aos métodos já descritos na literatura, tais como sequenciamento de um segmento do gene SSU‐rDNA, PCR‐kDNA e por RFLP (restriction fragment length polymorphisms) do gene hsp70.
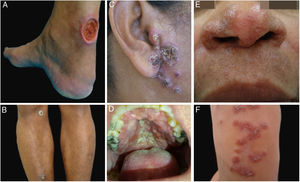
ResultadosNo período estudado foram atendidos 22 pacientes com suspeita diagnóstica de LTA. O grupo apresentava 12 pacientes do sexo masculino e 10 pacientes do feminino. A idade variou de 13 a 78 anos, com média de 45,72 ± 18,41 e mediana de 43,50 anos. O tempo de duração da lesão de pele variou de um mês a 108 meses, com média de 28,31 ± 35,49 e mediana de 10. A forma clínica mais encontrada foi úlcera de pele (13 pacientes), seguida por úlceras em mucosas (quatro pacientes com lesões nasais e dois com orais), lesões cutâneas papulosas (dois pacientes) e lesão nodular cutânea em um paciente (figs. 1 e 2).
Exemplos clínicos dos pacientes com lesões clínicas cuja pesquisa PCR‐HRM determinou a espécie de Leishmania sp. (A), paciente 11 (L. infantum chagasi); (B), paciente 13 (L. amazonensis + L. infantum chagasi); (C), Paciente 14 (L. amazonensis + L. infantum chagasi); (D), paciente 7 (L. brasiliensis); (E), paciente 3 (L. amazonensis); (F), paciente 5 (L. amazonensis).
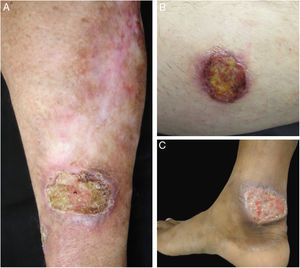
Exemplos dos pacientes com lesões clínicas cujas pesquisas PCR‐HRM não detectaram Leishmania sp. (A), paciente 17, com úlcera em membro superior; (B), paciente 21, com úlcera em membro inferior cuja biópsia evidenciou presença de amastigotas no tecido; (C), paciente 21 com úlcera em membro inferior.
As características epidemiológicas, clínicas, histopatológicas e pesquisa direta do parasita no tecido lesado dos 22 pacientes incluídos no estudo estão especificadas na tabela 1.
Aspectos clínicos, epidemiológicos e laboratoriais dos casos estudados
| Características das amostras | Característica das lesões | Evidência do agente | Testes complementares | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Paciente | Procedência (Estado) | Forma clínica | Tecido/local | Tempo de lesão (meses) | Exame direto | Histopatológico | PCR SSU | PCR | SSU | HSP70 | HSP70 |
| IHQ | kDNA | Seq. | RFLP | HRM | |||||||
| 1 | MG | Úlcera | Pele/membro | 4 | + | NR | + | + | NR | L. amazonensis | L. amazonensis |
| 2 | PR | Úlcera | Pele/membro | 4 | + | + | + | + | NR | L. amazonensis | NR |
| 3 | BA | Úlcera | Mucosa nasal | 72 | − | NR | + | + | NR | L. amazonensis | L. amazonensis |
| 4 | BA | Pápula | Pele/nariz | 24 | + | − | + | + | L. amazonensis | L. amazonensis | NR |
| 5 | SE | Nódulos | Pele/membro | 108 | − | NR | + | + | L. amazonensis | L. amazonensis | L. amazonensis |
| 6 | BA | Úlcera | Pele/orelha | 6 | + | NR | + | + | NR | NR | L. braziliensis |
| 7 | MG | Úlcera | Mucosa oral | 3 | NR | + | + | + | L. (Viannia) | NR | L. braziliensis |
| 8 | MG | Pápula | Pele/periocular | 18 | + | + | + | + | NR | NR | L. braziliensis |
| 9 | BA | Úlcera | Pele/membro | 12 | + | NR | + | NR | NR | NR | L. braziliensis |
| 10 | BA | Placa ulcerada | Pele/membro | 12 | NR | − | + | − | NR | NR | L. guyanensis |
| 11 | AL | Úlcera | Pele/membro | 96 | − | NR | + | − | NR | NR | L. guyanensis |
| 12 | BA | Úlcera | Pele/membro | 2 | + | NR | + | + | L. i. chagasi | L. i. chagasi | L. i. chagasi |
| 13a | RN | Placa ulcerada | Pele/membro | 7 | NR | + | + | + | L. amazonensis/ L. i. chagasi | L. amazonensis/ L. i. chagasi | L. amazonensis/ L. i. chagasi |
| 14& | BA | Úlcera | Pele/orelha | 5 | + | NR | + | + | L. amazonensis/ L. i .chagasi | L. amazonensis/ L. i. chagasi | L. amazonensis/ L. i. chagasi |
| 15 | SP | Úlcera | Pele/membro | 5 | NR | − | − | NR | NR | NR | NR |
| 16 | BA | Úlcera | Mucosa oral | 12 | − | NR | − | NR | NR | NR | NR |
| 17 | BA | Úlcera | Pele/membro | 8 | − | NR | − | NR | NR | NR | NR |
| 18 | BA | Úlcera | Mucosa nasal | 60 | − | NR | − | NR | NR | NR | NR |
| 19 | MG | Úlcera | Pele/membro | 4 | − | NR | − | NR | NR | NR | NR |
| 20 | BA | Úlcera | Mucosa nasal | 60 | − | NR | − | NR | NR | NR | NR |
| 21 | SP | Úlcera | Pele/membro | 5 | NR | + | − | NR | NR | NR | NR |
| 22 | SP | Úlcera | Mucosa nasal | 96 | − | NR | − | NR | NR | NR | NR |
NR, não realizado; e testes moleculares aplicados em duas amostras distintas, das quais uma indicou a presença de L. amazonensis e outra, L. infantum chagasi.
Para diagnóstico, foram feitos esfregaços em lesões de 17 pacientes (oito positivas) e biópsias de pele em todos os 22 pacientes (14 positivas), com análise de todas as amostras pela coloração de rotina (hematoxilina‐eosina) e feitura de estudo imuno‐histoquímico para detecção de Leishmania em oito pacientes. Em 10 pacientes foi feita mais de uma biópsia com o intuito de diagnosticar a doença. Ao todo, foram 38 blocos analisados por histologia, PCR‐SSU, PCR‐kDNA, SSU‐Seq, PCR hsp70‐RFLP, PCR hsp70‐HRM (tabela 1).
Onze pacientes apresentaram formas amastigotas positivas em exame de esfregaço e/ou exame histológico, dos quais apenas dois pacientes foram positivos nos dois testes. Em 11 pacientes não foram encontradas formas amastigotas, por um ou outro teste (tabela 1).
Dos 22 pacientes com suspeita de LTA atendidos no período, 14 tiveram a espécie do parasita identificada por SSU‐Seq, PCR hsp70‐RFLP ou PCR hsp70‐HRM: cinco casos (35,6%) de infecções por L. amazonensis, quatro (28,5%) por L. braziliensis, dois (14,4%) por L. amazonensis + L. infantum chagasi, dois (14,4%) por L. guyanensis e um (7,1%) por L. infantum chagasi. Interessante notar que o sequenciamento do fragmento SSU não permite a identificação das espécies do subgênero L. (Viannia), mas houve coincidência da espécie identificada, ou dentro desse subgênero, ou nos casos em que houve uma dupla identificação de L. amazonensis + L. infantum chagasi (tabela 1).
Das amostras de 11 pacientes em que foram detectadas formas amastigotas por meio da pesquisa direta em esfregaço de lesão e/ou exame histopatológico de fragmento de pele, foi possível identificar a espécie do parasita em 10 delas e somente uma foi negativa pelos métodos moleculares empregados. Em contrapartida, das 11 amostras negativas pelos exames de esfregaço e/ou exames histopatológicos, houve identificação do protozoário em quatro (tabela 2). Interessante observar que um dentre esses quatro pacientes, o paciente 5, apresentava lesões cutâneas nodulares havia 108 meses, de difícil diagnóstico clínico e patológico (fig. 1 e tabela 1).
Resultado da evidência do parasita pela pesquisa direta em esfregaço de lesão e/ou exame histopatológico e identificação da Leishmania sp. pelo método de PCR‐HRM correlacionando com a forma clínica
| Lesão cutaneomucosa | Presença de parasita (ED/HP) | Ausência de parasita (ED/HP) | Total | ||
|---|---|---|---|---|---|
| PCR‐HRM positivo | PCR‐HRM negativo | PCR‐HRM positivo | PCR‐HRM negativo | ||
| Pele | |||||
| Úlcera em pele | 7 | 1 | 2 | 3 | 13 |
| Pápulas | 2 | 0 | 0 | 0 | 2 |
| Nódulos | 0 | 0 | 1 | 0 | 1 |
| Mucosa | |||||
| Úlcera oral | 1 | 0 | 0 | 1 | 2 |
| Úlcera nasal | 0 | 0 | 1 | 3 | 4 |
| Total | 10 | 1 | 4 | 7 | 22 |
ED, pesquisa em exame direto; HP, exame histopatológico.
O PCR‐SSU foi positivo para 14 dos pacientes; a espécie foi identificada em todos, ou por SSU‐Seq ou PCR hsp70‐RFLP ou PCR hsp70‐HRM. Ainda, no DNA desses 14 pacientes foi aplicado o teste PCR‐kDNA, que resultou em 12 amostras positivas.
DiscussãoAinda hoje há dificuldade de fazer o diagnóstico preciso em pacientes com suspeita de leishmaniose cutânea e/ou mucosa nos casos em que não são encontrados parasitas nas lesões. São solicitados vários exames complementares para tentar chegar ao diagnóstico e excluir outras doenças como micobacterioses, sarcoidose, linfomas, micoses profundas, entre outras.1,6,8 Dessa maneira, é importante que se possa dispor de um exame rápido para diagnóstico, com especificidade e alta sensibilidade, o que evitaria exames e consultas excessivos, além de provas terapêuticas com efeitos colaterais indesejáveis. A introdução da técnica de PCR para demonstração do parasita tem evoluído muito nos últimos anos; a técnica acoplada com HRM, que tem como alvo o gene hsp70 de Leishmania, possibilita a identificação laboratorial de todas as espécies com especificidade e alta sensibilidade.34,37,38
Além disso, os resultados apresentados ressaltam o fato de que, mesmo em se tratando de um hospital público terciário de referência na maior cidade do país, o acesso a exames diagnósticos simples não é sempre disponível. Dos 22 casos estudados, houve a possibilidade de se fazer o estudo imuno‐histoquímico para Leishmania em apenas oito (36,3%). A pesquisa direta do parasita no esfregaço da lesão corada com Giemsa foi o exame mais acessível, feito em 17 pacientes (77,2%).
A demonstração da presença do parasita pela detecção de seu DNA se dá por testes considerados potencialmente sensíveis, uma vez que a positividade é resultado da amplificação exponencial do alvo por PCR. De fato, os testes de PCR aplicados em 14 dos 22 pacientes foram positivos com SSU (63,64%) e 12 foram positivos com kDNA (54,55%) – esses também foram positivos para o PCR‐SSU. No entanto, esses alvos não permitem a discriminação da espécie do parasita.
Houve concordância na identificação da espécie nos testes SSU‐Seq, hsp70‐RFLP e hsp70‐HRM, mas ressaltamos que os dois primeiros são testes trabalhosos, que requerem a manipulação do produto de PCR, além de um técnico treinado para executar e analisar os resultados.
Em um paciente em que foi detectada a presença de amastigotas por histopatologia, os testes PCR‐SSU e hsp70‐HRM foram negativos. Uma possível falha na detecção de DNA do parasita pode ter explicações biológicas ou físico‐químicas. A primeira baseia‐se no fato de que lesões cutâneas e mucocutâneas causadas principalmente por L. braziliensis têm como característica comum a escassez ou ausência de parasitas, dificultam o diagnóstico laboratorial. A segunda é baseada no fato de que amostras biológicas que passam por tratamentos de fixação para análises histológicas estão sujeitas à degradação de DNA ou à inibição da enzima polimerase, principalmente em função de sua exposição à formalina ou a reagentes usados na preparação das amostras.1,3,6,38 No caso do paciente com evidência de formas amastigotas pelo exame de imuno‐histoquímica, a amostra foi submetida a um teste de amplificação do DNA do hospedeiro por PCR convencional e teve o alvo amplificado. Esse teste é feito como rotina para verificar a qualidade do DNA e a interferência da possível presença de inibidores de polimerase nas amostras. Esse resultado sugere, então, que a ausência ou extrema escassez de parasitas nessa amostra pode ser uma explicação convincente para a negatividade do teste de detecção e identificação do parasita.
A identificação de L. amazonensis em lesões de cinco pacientes oriundos da Bahia (dois), do Sergipe (um), do Paraná (um) e de Minas Gerais (um) inicialmente nos surpreendeu, visto que nesses estados L. braziliensis é a espécie mais comumente relacionada aos casos de leishmaniose cutaneomucosa. L. amazonensis é classicamente considerada restrita a áreas da Amazônia, com transmissão por vetores silvestres e tem como reservatórios principalmente roedores. No entanto, recentemente têm sido descritas mudanças no perfil epidemiológico dessa espécie. Na Bahia, onde é mais comum a presença de L. braziliensis, já em 1988 foi demonstrada, por meio de painéis de anticorpos monoclonais, a presença de L. amazonensis que causou lesões cutâneas, e mais raramente, lesões mucosas.39 Em outras regiões como Mato Grosso do Sul, Santa Catarina, Paraná e Rio de Janeiro foram identificados casos humanos isolados de leishmaniose tegumentar por L. amazonensis.40 Essa espécie também foi identificada em cães no estado de São Paulo41,42 e mais recentemente em áreas endêmicas do Sudeste do Brasil.43,44 Assim, consideramos que existe uma possibilidade de que uma proporção desconhecida de casos de leishmaniose cutânea esteja sendo causada por L. amazonensis fora da Amazônia. Mais estudos epidemiológicos são necessários já que são descritos indícios de que o perfil de transmissão da leishmaniose tegumentar pode estar mudando.
Também foi inesperado o achado de lesão mucosa causada por L. amazonensis. Raros casos humanos foram descritos na literatura; entretanto, já foi reproduzida experimentalmente em ratos.45 A identificação de L. guyanensis em dois pacientes oriundos do Nordeste do Brasil também é outro dado inesperado, visto que essa espécie tem ciclo de transmissão aparentemente limitado à Região Norte.46,47 Porém, vale ressaltar que a origem geográfica referida nos prontuários não aponta onde os pacientes foram infectados, desde que há uma movimentação nas diversas regiões do país, e informações mais precisas nesse sentido muitas vezes são escassas em estudos retrospectivos. Por outro lado, a ocorrência dessas espécies fora das áreas endêmicas alerta para a possibilidade de uma nova epidemiologia da doença. Não sabemos se está ocorrendo uma ampliação de sua distribuição geográfica ou se essas espécies já estavam sendo distribuídas no território nacional havia tempos e modificações ambientais estariam aumentando a exposição de animais como cães e homens a parasitas em áreas onde eles não haviam sido descritos. Novos métodos de identificação da espécie dos parasitas permitem que sua distribuição geográfica seja revista, contribuem para o entendimento da epidemiologia da doença.
A identificação de um caso com L. infantum chagasi é considerada uma raridade na LTA no Brasil, visto que essa espécie é geralmente relacionada ao acometimento visceral da doença.4,5 Casos isolados foram descritos em associação com infecção por HIV, em pacientes jovens, e até mesmo um em área urbana do Rio de Janeiro, sem qualquer comorbidade associada,5 como o caso aqui apresentado. Infecções mistas causadas por mais de uma espécie de Leishmania, ou até mesmo com tripanossomatídeos de outros gêneros parasitando um mesmo hospedeiro, apesar de raras, têm sido descritas na literatura há alguns anos.48 Martinez et al., em 2002, descreveu o primeiro caso de detecção de L. amazonensis e L. infantum chagasi em uma mesma lesão em paciente boliviano, situação semelhante à identificada em caso aqui apresentado.49 Essas duas espécies foram encontradas em outro paciente, mas em amostras distintas, o que não nos permite inferir se foram duas infecções temporalmente distintas ou uma dupla infecção inicial.
Este estudo apresenta limitações pelo fato de ser retrospectivo e pelo pequeno número de pacientes, mas pode apontar, pela ampliação das observações para mais casos, para novos paradigmas na identificação de espécies de Leishmania relacionadas às diversas áreas geográficas e às formas de doença. As amostras de DNA de pacientes com diagnóstico final negativo para leishmaniose também devem ser consideradas informativas. Houve concordância entre os resultados negativos e a ausência de formas amastigostas em 19 blocos analisados de oito pacientes. Em termos práticos, um resultado negativo confiável pode evitar a coleta de mais amostras, por falha de diagnóstico além de poupar um desnecessário tratamento quimioterápico.
Idealmente, a feitura de exame de PCR para detecção e identificação de Leishmania deve ser acessível à maioria dos serviços de saúde de atendimento aos doentes no Brasil. Anteriormente à elaboração deste estudo e ao acesso à identificação das espécies por hsp70‐HRM nos blocos de parafina, os pacientes atendidos em nosso serviço foram tratados com base nos dados clínicos (presença de lesão mucocutânea), dados epidemiológicos e exames complementares, quando disponíveis. Nota‐se nos casos aqui descritos que o acesso a exames complementares não é rotina e naqueles com ausência de formas amastigotas há dificuldade para confirmação diagnóstica; dessa forma, muitas vezes é indicada a feitura de prova terapêutica com possíveis efeitos colaterais, o que muitas vezes gera desconforto e sofrimento ao paciente.
A identificação das espécies de Leishmania é importante para a escolha de tratamento.1,3,47 Sabe‐se que a L. braziliensis pode levar a comprometimento de mucosas, mesmo em pacientes que tiveram uma única úlcera,3,6 e há possibilidade de casos de acometimento visceral em pacientes com lesões cutâneas causadas por L. infantum chagasi.4,5,50 Assim, nesses casos, deve‐se indicar tratamento sistêmico em vez de locais, como as infiltrações intralesionais com antimoniato de meglumina, preconizadas para tratamento de lesões ulceradas únicas na pele.3
ConclusãoO diagnóstico da leishmaniose está entre os maiores desafios no que se refere ao combate à doença. A identificação correta da espécie de Leishmania em amostras parafinadas de pele e mucosa é fonte de dados importantes para estudos ecológicos, epidemiológicos e sobre a biologia do parasita e sua interação com o hospedeiro e pode ser indicativa do delineamento do protocolo terapêutico.
Dos três testes empregados na identificação da espécie, dois são muito trabalhosos e requerem técnico treinado para a interpretação dos resultados. O fato de o resultado hsp70‐HRM ser fornecido por uma máquina, além de tornar possível sua automação, elimina potenciais contaminações decorrentes de manipulações necessárias para a análise de resultados obtidos por PCR convencional, pois essa técnica é baseada em análises do produto de PCR em tubos lacrados, sem a necessidade de manipulação.
A elaboração deste trabalho tornou possível a identificação de espécies do parasita em amostras clínicas em 10/11 pacientes com formas amastigotas positivas. Além disso, demonstrou a presença de Leishmania em 4/11 pacientes nos quais não haviam sido encontradas formas amastigotas nas amostradas estudadas e definitivamente afastou o diagnóstico de leishmaniose em 7/11 pacientes com ausência de parasitas nos exames diretos.
Além de L. braziliensis, L. guyanensis, L. amazonensis e L. infantum chagasi, foi detectada associação das últimas duas espécies. Na prática, resultados como esse indicam que a ocorrência de casos tidos como raros podem ser mais comuns do que previamente descritos.
Consideramos que estudos prospectivos com populações maiores devem ser feitos e também sugerimos aplicar a técnica hsp70‐HRM em material obtido de esfregaços, o que possibilita estudos em regiões remotas do país.
Suporte financeiroNenhum.
Contribuição dos autoresJohn Verrinder Veasey: Análise estatística; aprovação da versão final do manuscrito; concepção e planejamento do estudo; elaboração e redação do manuscrito; obtenção, análise e interpretação dos dados; participação efetiva na orientação da pesquisa; participação intelectual em conduta propedêutica e/ou terapêutica de casos estudados; revisão crítica da literatura; revisão crítica do manuscrito.
Ricardo Andrade Zampieri: Aprovação da versão final do manuscrito; concepção e planejamento do estudo; elaboração e redação do manuscrito; obtenção, análise e interpretação dos dados; participação efetiva na orientação da pesquisa; revisão crítica da literatura; revisão crítica do manuscrito.
Rute Facchini Lellis: Obtenção, análise e interpretação dos dados; participação intelectual em conduta propedêutica e/ou terapêutica de casos estudados.
Thaís Helena Proença de Freitas: Análise estatística; aprovação da versão final do manuscrito; concepção e planejamento do estudo; elaboração e redação do manuscrito; obtenção, análise e interpretação dos dados; participação efetiva na orientação da pesquisa; participação intelectual em conduta propedêutica e/ou terapêutica de casos estudados; revisão crítica da literatura; revisão crítica do manuscrito.
Lucile Maria Floeter Winter: Análise estatística; aprovação da versão final do manuscrito; concepção e planejamento do estudo; elaboração e redação do manuscrito; obtenção, análise e interpretação dos dados; participação efetiva na orientação da pesquisa; revisão crítica da literatura; revisão crítica do manuscrito.
Conflitos de interesseNenhum.
Como citar este artigo: Veasey JV, Zampieri RA, Lellis RF, Freitas THP, Winter LMF. Identification of Leishmania species by high‐resolution DNA dissociation in cases of American cutaneous leishmaniasis. An Bras Dermatol. 2020;95:459–68.
Trabalho realizado na Clínica de Dermatologia, Hospital da Santa Casa de Misericórdia de São Paulo, Santa Casa de Misericórdia de São Paulo, São Paulo, SP, Brasil.